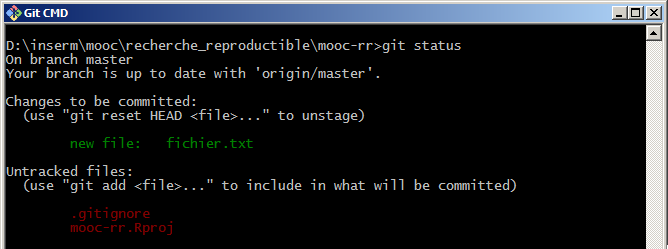
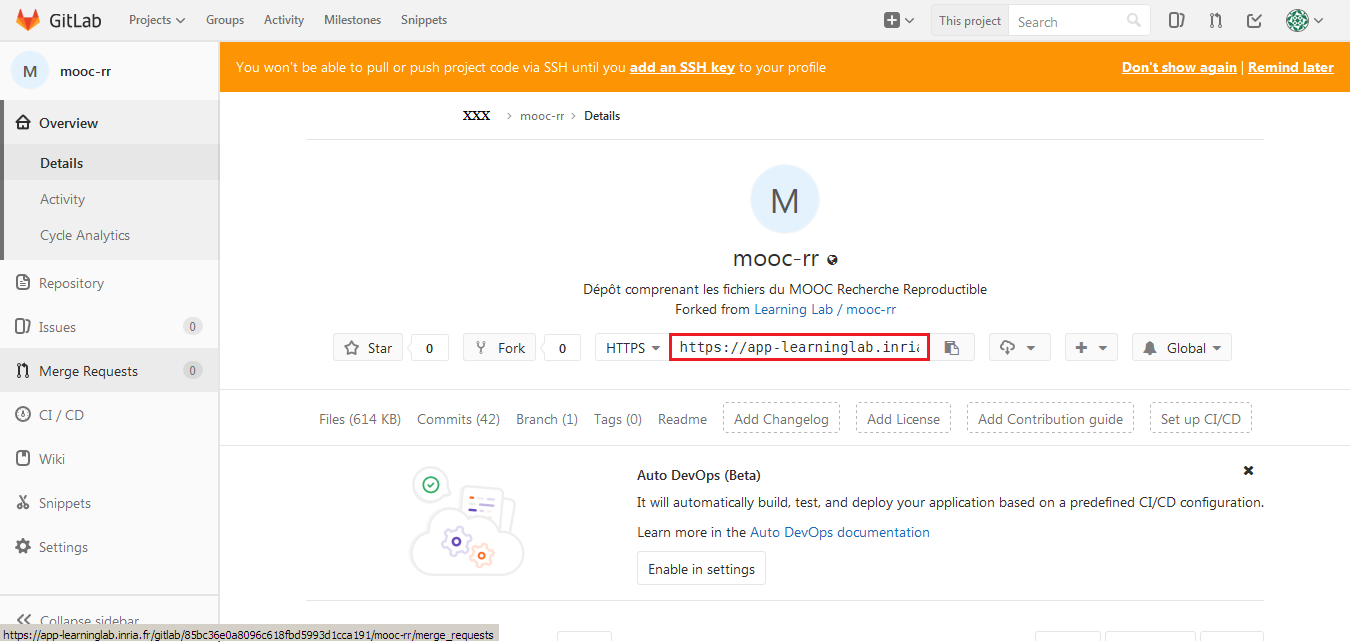
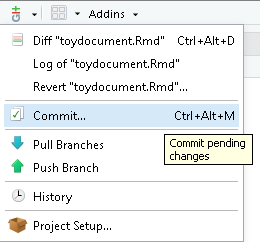
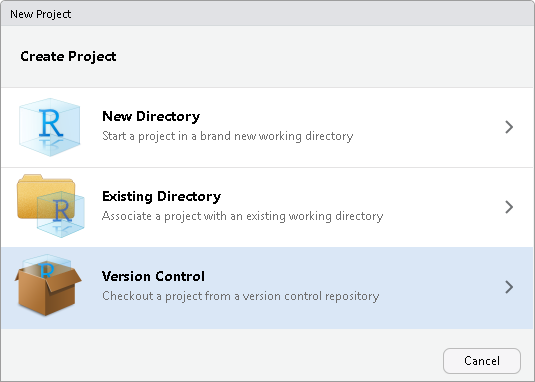
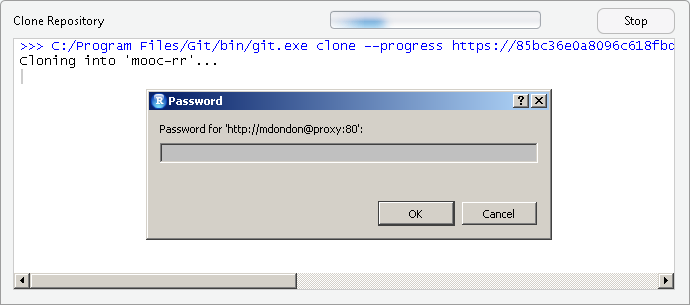
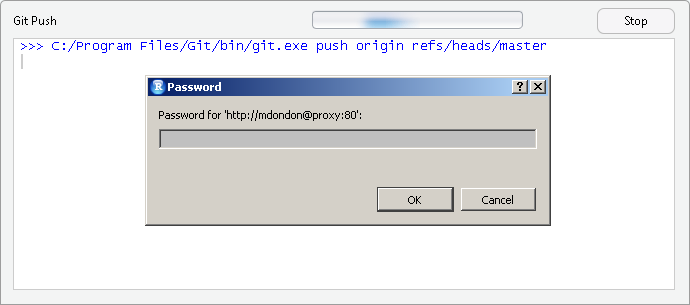
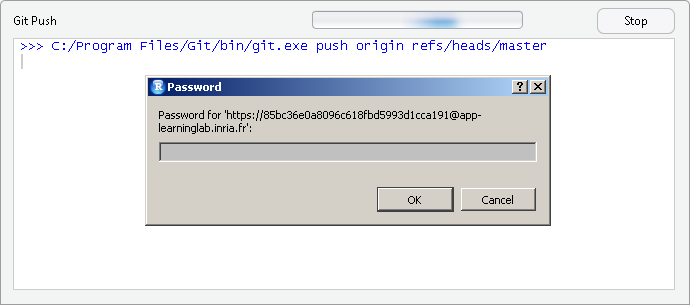
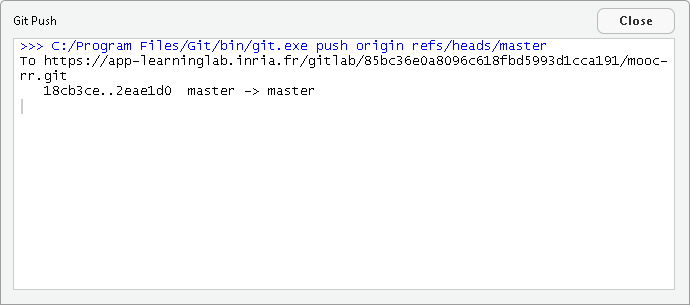
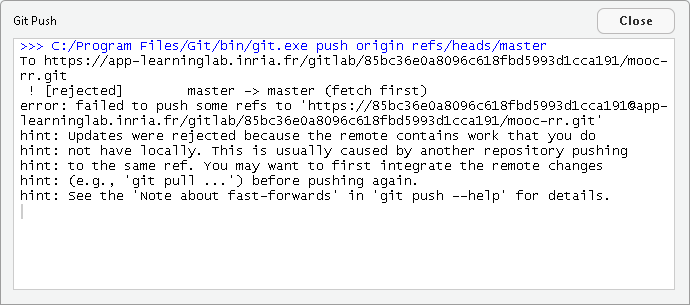
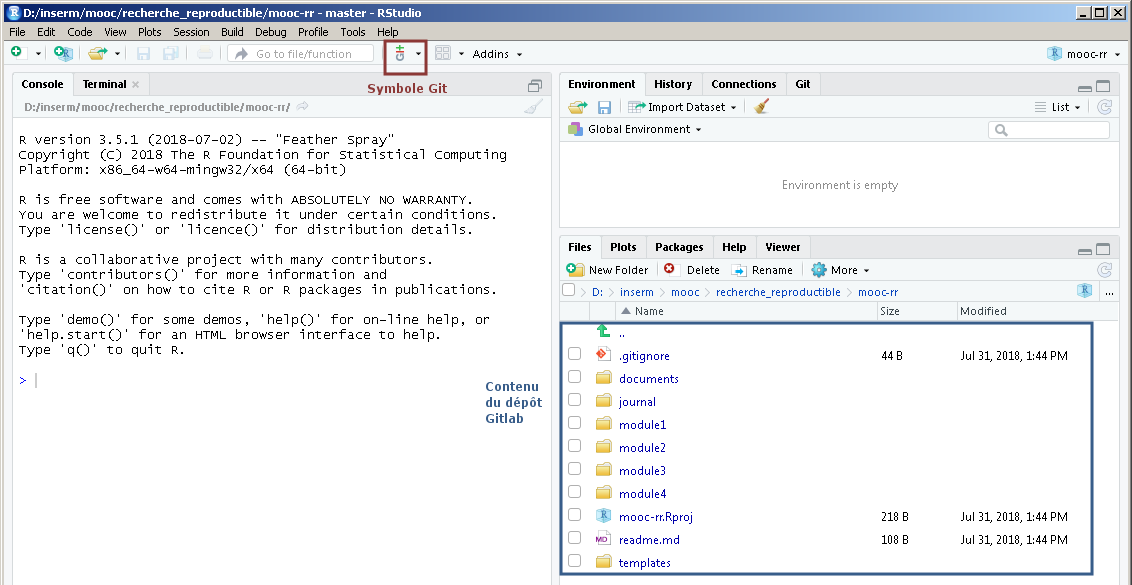
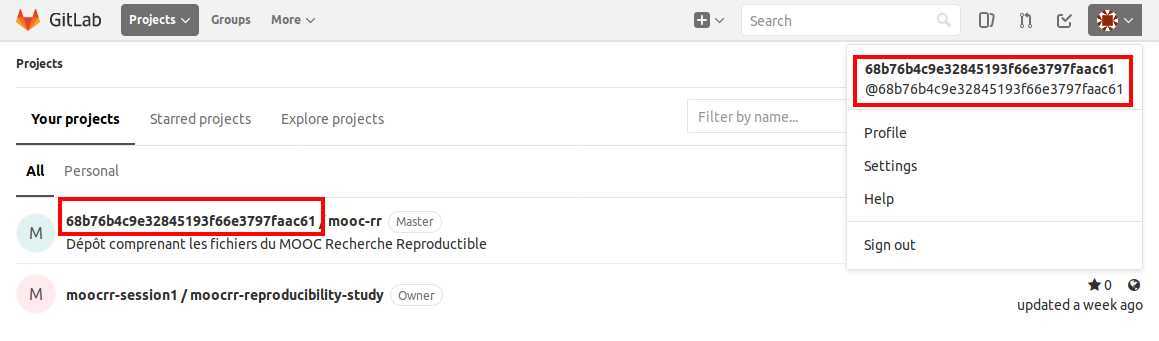
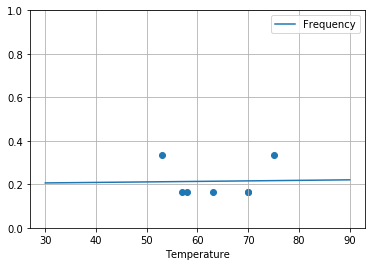
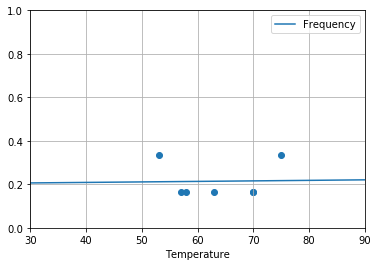
Commits (435)
- 335 additional commits have been omitted to prevent performance issues.
Showing
.gitlab-ci.yml
0 → 100644
LICENSE
0 → 100644
Makefile
0 → 100644
Makefile.ressources
0 → 100644
Readme.md
0 → 100644
bin/pandoc_fixer.pl
0 → 100755
File moved
documents/notebooks/cars.csv
0 → 100644
File moved
17.9 KB
This source diff could not be displayed because it is too large. You can view the blob instead.
File added
File added
File added
16.9 KB
27 KB
11.4 KB
51.9 KB
9.54 KB
8.02 KB
51.4 KB
70.1 KB
10.7 KB
5.04 KB
12.9 KB
17.5 KB
13.9 KB
51.9 KB
16.1 KB
7.43 KB
21.7 KB
9.54 KB
8.95 KB
5.37 KB
9.64 KB
23.7 KB
10.9 KB
10.1 KB
6.22 KB
14.9 KB
14.9 KB
45.7 KB
gitlab-ci.org
0 → 100644
html_src_files.lst
0 → 100644
86.8 KB
92.7 KB
98.6 KB
File moved
30.9 KB
File moved
File moved
File moved
File moved
27.7 KB
File added
module1/slides/gitdags.sty
0 → 100644
52.5 KB
47.6 KB
File added
File added
14.7 KB
50.5 KB
25.8 KB
150 KB
6.19 KB
File added
File added
67.1 KB
183 KB
File added
This source diff could not be displayed because it is too large. You can view the blob instead.
This source diff could not be displayed because it is too large. You can view the blob instead.
File added
This source diff could not be displayed because it is too large. You can view the blob instead.
File added
This source diff could not be displayed because it is too large. You can view the blob instead.
File added
This source diff could not be displayed because it is too large. You can view the blob instead.
File added
This source diff could not be displayed because it is too large. You can view the blob instead.
160 KB
81.8 KB
This source diff could not be displayed because it is too large. You can view the blob instead.
This source diff could not be displayed because it is too large. You can view the blob instead.
File added
File added
No preview for this file type
No preview for this file type
131 KB
56.8 KB
104 KB
149 KB
143 KB
49.2 KB
File moved
File moved
File moved
File moved
File moved
File moved
File moved
File moved
File moved
File moved
File moved
File moved
File moved
File moved
This source diff could not be displayed because it is too large. You can view the blob instead.
File moved
File moved
File moved
File moved
File moved
File moved
File moved
File moved
No preview for this file type
No preview for this file type
No preview for this file type
This source diff could not be displayed because it is too large. You can view the blob instead.
File moved
File moved
File moved
File moved
File moved
module3/ressources/Makefile
0 → 100644
16 KB
21 KB
26.7 KB
44.6 KB
This source diff could not be displayed because it is too large. You can view the blob instead.
This source diff could not be displayed because it is too large. You can view the blob instead.
5.15 KB
6.94 KB
7.02 KB
6.86 KB
This source diff could not be displayed because it is too large. You can view the blob instead.
public/readtheorg.css
0 → 100644