relecture and final adjustments
Showing
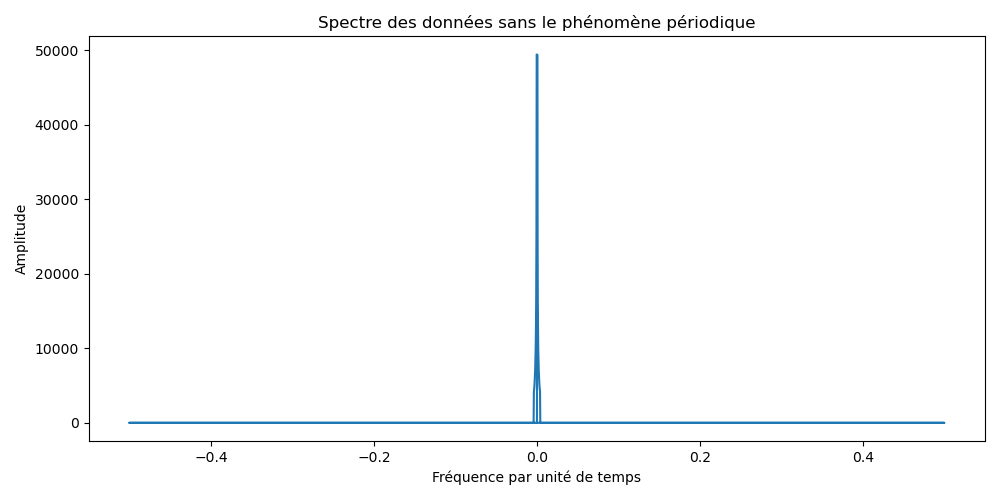
24.7 KB | W: | H:
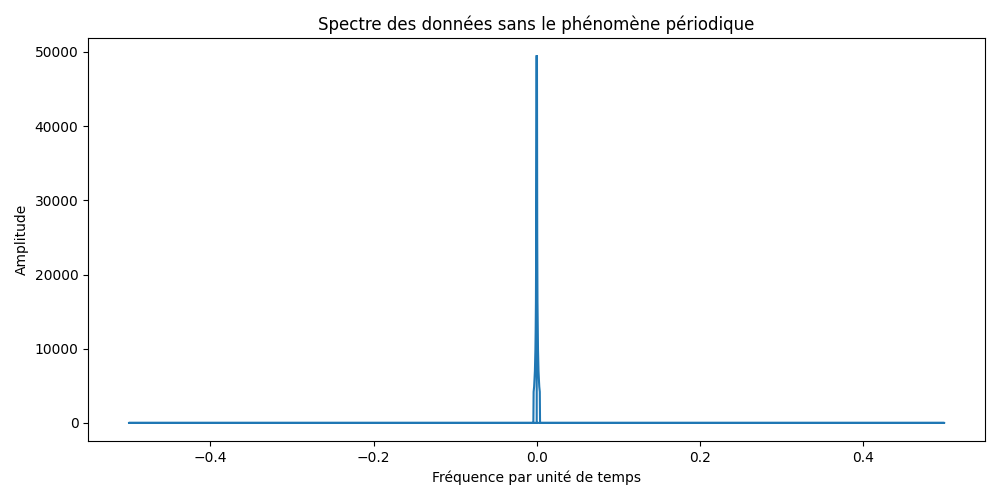
24.9 KB | W: | H:




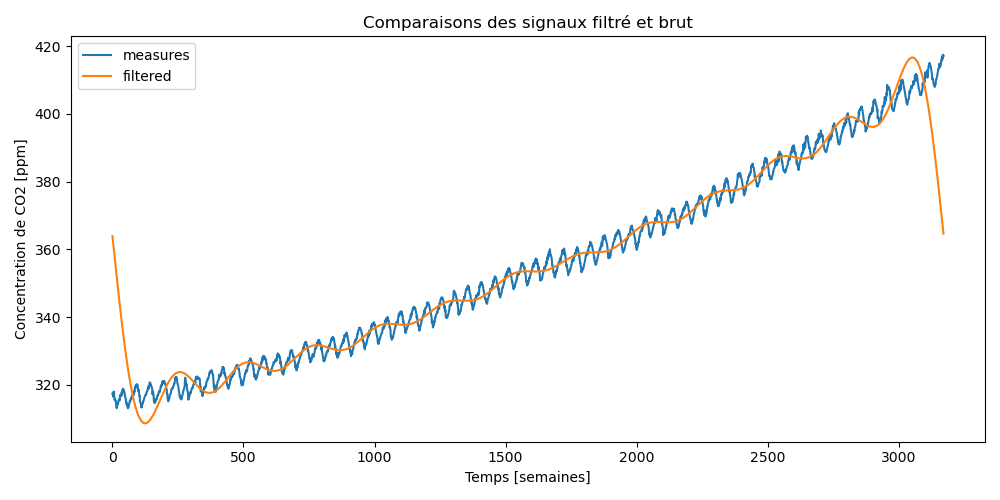
65.3 KB | W: | H:
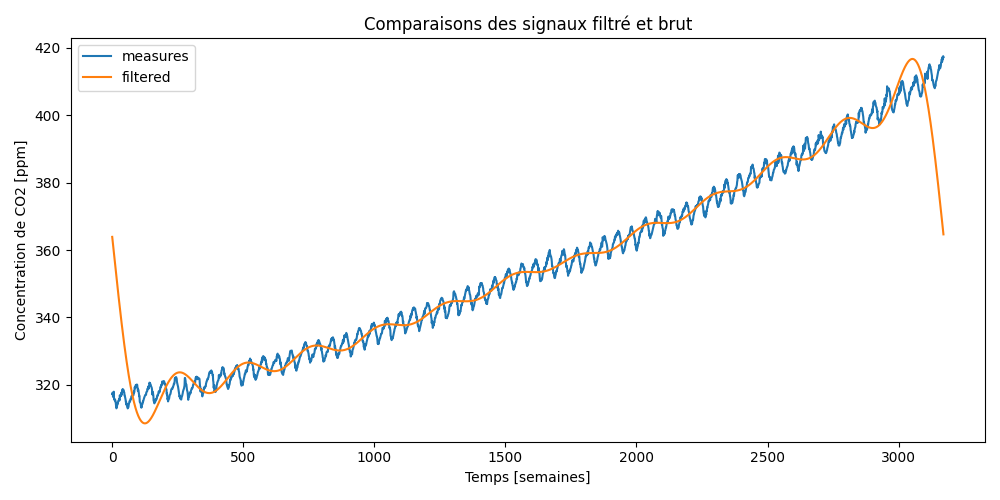
65.8 KB | W: | H:




41.9 KB | W: | H:
42.8 KB | W: | H:





45.7 KB | W: | H:
46.3 KB | W: | H:




25.8 KB | W: | H:

24.7 KB | W: | H:




41.3 KB | W: | H:
40.8 KB | W: | H:





65.6 KB | W: | H:

66.1 KB | W: | H:




42.9 KB | W: | H:

43.4 KB | W: | H:




39.4 KB | W: | H:

39.7 KB | W: | H:




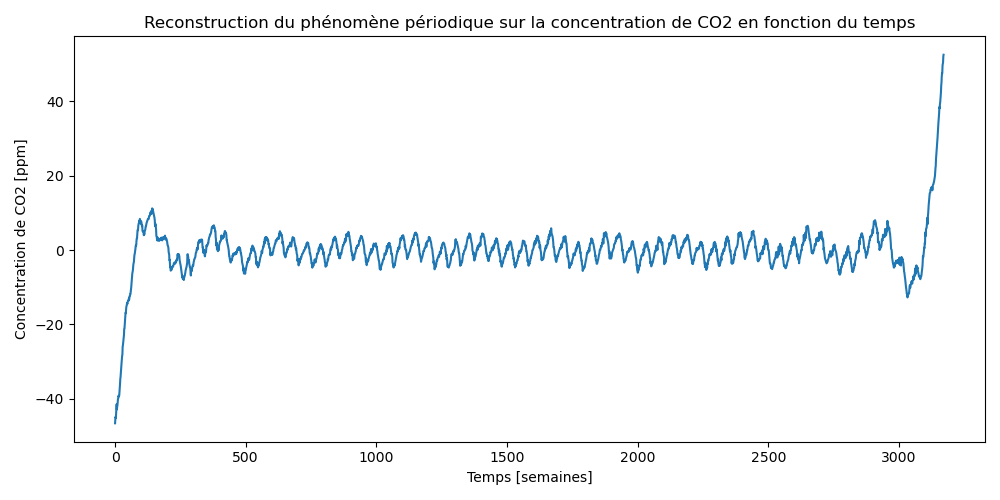
53.3 KB | W: | H:
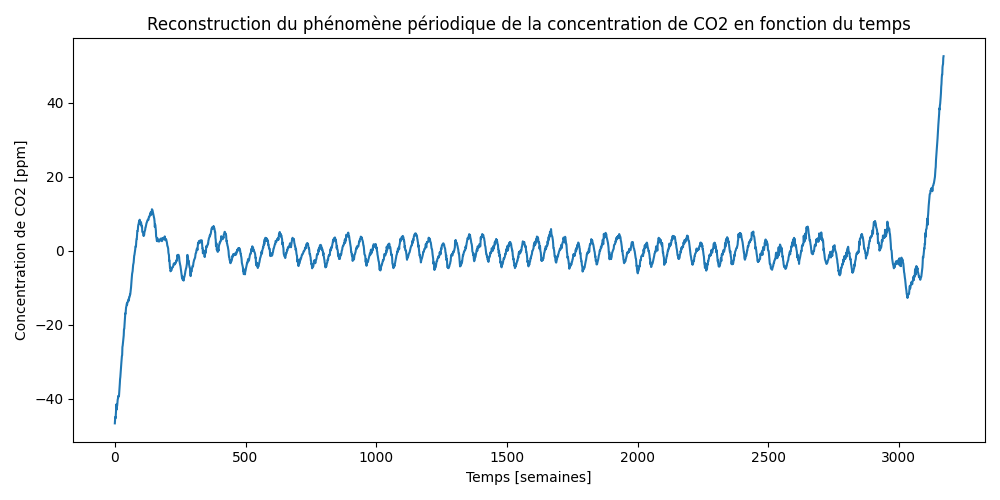
53.5 KB | W: | H:




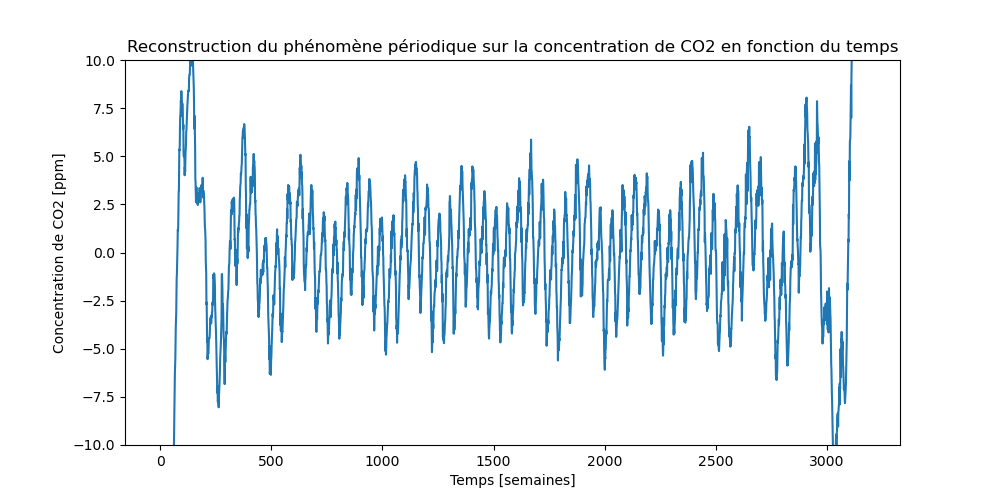
96.3 KB | W: | H:
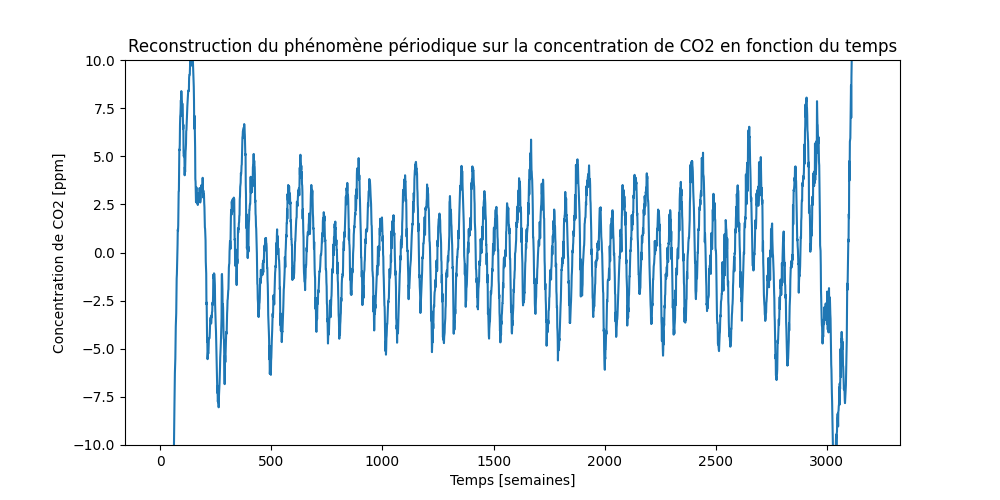
97.1 KB | W: | H:




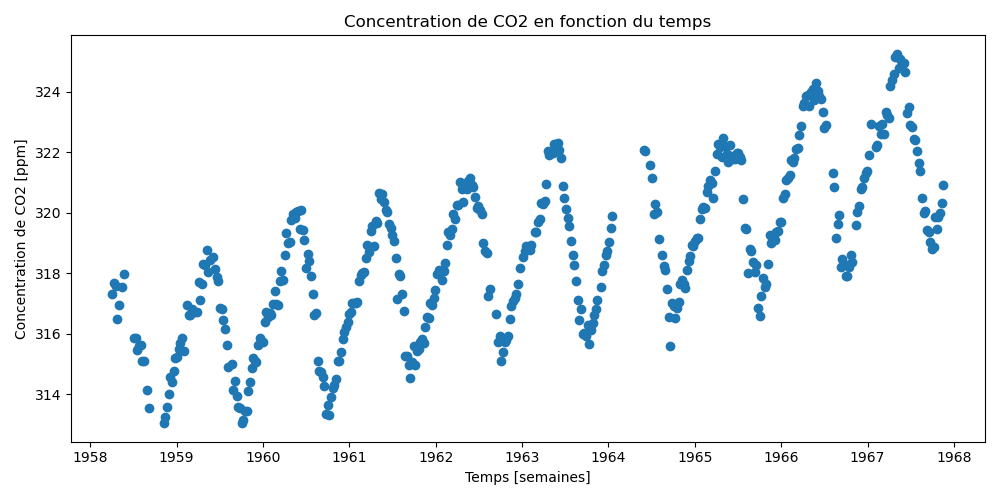
47.5 KB | W: | H:
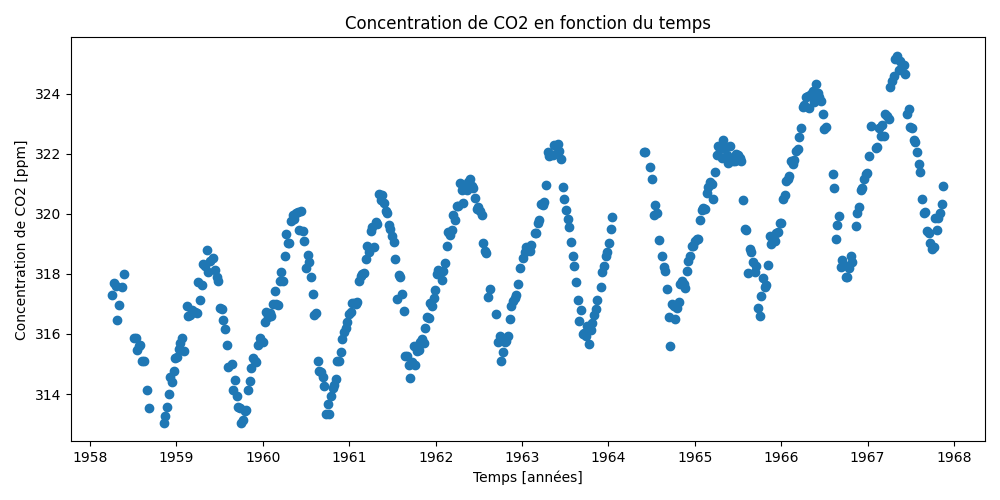
47.8 KB | W: | H:



