Merge branch 'master' of...
Merge branch 'master' of https://app-learninglab.inria.fr/moocrr/gitlab/3e6bf7b47a05a05ae3d6af86121dcb5d/mooc-rr
Showing
module2/exo1/montecarlo.png
0 → 100644
79.4 KB
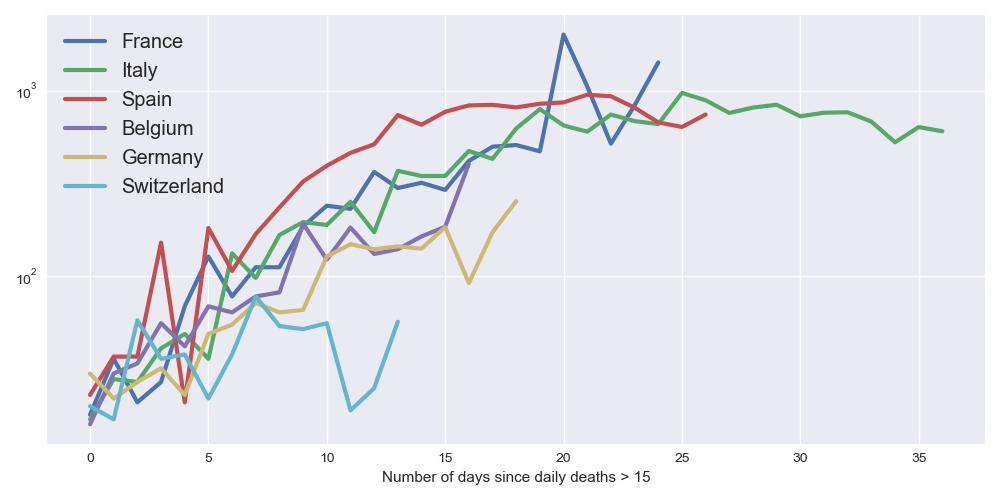
module2/exo4/daily.png
0 → 100644
82.8 KB
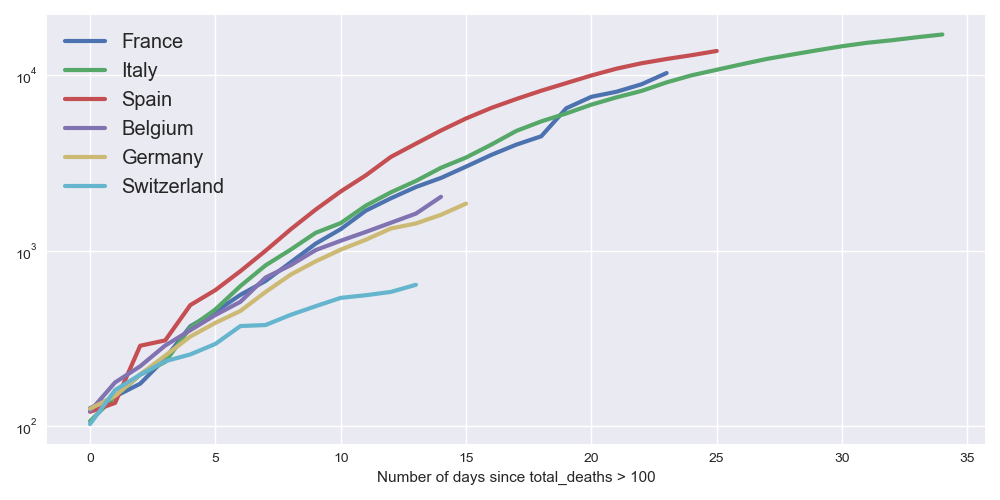
module2/exo4/total.png
0 → 100644
60.6 KB